近日,信息管理学院黄水清教授课题组研制完成后生动物基因表达和可变剪接数据分析平台(https://bioinfo.njau.edu.cn/metaExp),成果“MetazExp: a database for gene expression and alternative splicing profiles and their analyses based on 53 615 public RNA-seq samples in 72 metazoan species”在生物信息学领域权威期刊Nucleic Acids Research(IF=16.9)在线发表。

高通量RNA-seq测序技术广泛应用于生物学研究,产生了海量转录组测序数据。重分析、共分析来自不同课题产生的原始数据可获得更多生物学假设。然而,复杂的数据处理要求和巨大的数据规模加剧了数据利用难度,造成海量数据难于重利用的尴尬局面。
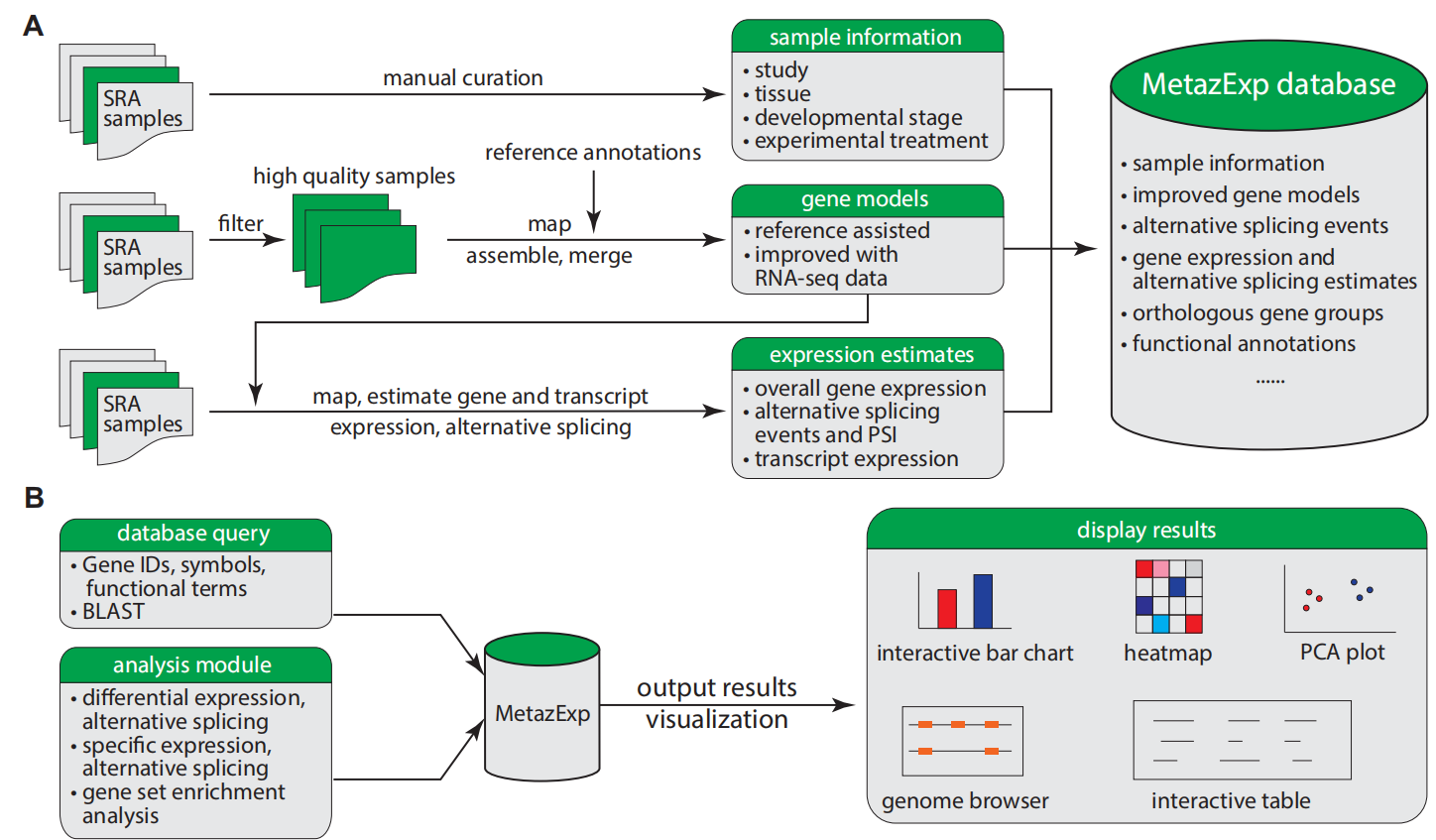
为消除生物学研究人员和大数据之间的沟渠,提升公共数据的利用率,该团队将情报学的数据采集、信息组织、领域知识挖掘方法应用于生物信息领域。以72种后生动物为对象,全面搜集整理了对应的53 615个测序数据,对样品品系、基因型、组织、发育阶段、处理条件等重要生物学属性进行了标注,开发pipeline优化基因模型、识别可变剪接事件、完善基因组注释,并对测序样本中基因表达和可变剪接水平分别在多个层次上进行定量。在此基础上,开发了具有检索和在线分析功能的平台,可自动生成各种实用图表,以便科研人员获得有意义的生物学见解,为深入研究相关基因功能提供有价值的参考。
南京农业大学为论文的第一作者和共同通信作者单位。我校信息管理学院刘金定副教授为论文第一作者,刘金定副教授、黄水清教授和密西根州立大学黄温助理教授为论文共同通信作者。信息管理学院硕士研究生尹斐、博士研究生郎坤、前沿交叉研究院段荣静老师等参与了相关研究工作。
据了解,黄水清教授团队计划进一步拓展该方向的研究工作,构建全面覆盖植物、真菌、家畜、水产等农业生命科学对象领域的高通量RNA-seq数据分析平台。
原文链接为:https://academic.oup.com/nar/advance-article/doi/10.1093/nar/gkab933/6413608?searchresult=1。
本新闻内容转载自:http://news.njau.edu.cn/2021/1029/c18a116219/page.htm。